微軟已將通用擴散模型框架EvoDiff開源,結合了能夠處理大量且多元資料的特性,以及擴散模型特殊的控制能力,在序列空間中能以可控制的方法生成蛋白質。使用EvoDiff所訓練的模型,能夠生成多樣化且結構合理的蛋白質,特別的是,EvoDiff模型還可以生成以結構為基礎的模型所無法生成的模型,這代表使用序列為基礎的方法具有通用性。
蛋白質是重要的生物分子,其參與許多細胞生理過程(Cellular Process),像是鐵血紅蛋白能夠在血液中運送氧氣,胰島素能夠調節血糖水平,因此科學家對生成新的蛋白質很感興趣,無論是在工業還是醫療用途都很有價值。
目前主要用於生成蛋白質結構的是以結構為基礎的蛋白質設計模型,由於這類模型需要使用已知的蛋白質三維結構資料進行訓練,但是目前已知的蛋白質結構資料有限,因此限制了訓練資料集的範圍也就限制結構蛋白質生成模型的蛋白質設計空間。況且,有些蛋白質並不會形成固定的摺疊結構,因此這類生成模型可以設計的蛋白質也就缺失了一大類。
微軟研究人員提出的EvoDiff框架,是結合進化(Evolutionary)與擴散(Diffusion)兩個方法,使用進化規模的蛋白質資料,並用這些資料建立一種擴散模型。所謂進化規模,指的是涵蓋大規模進化歷史,足以反映蛋白質長時間進化的資料,這類資料可以讓模型在探索和設計新蛋白質時表現得更好。考慮蛋白質三維結構取得困難,研究人員選擇讓EvoDiff模型在蛋白質序列空間進行生成。
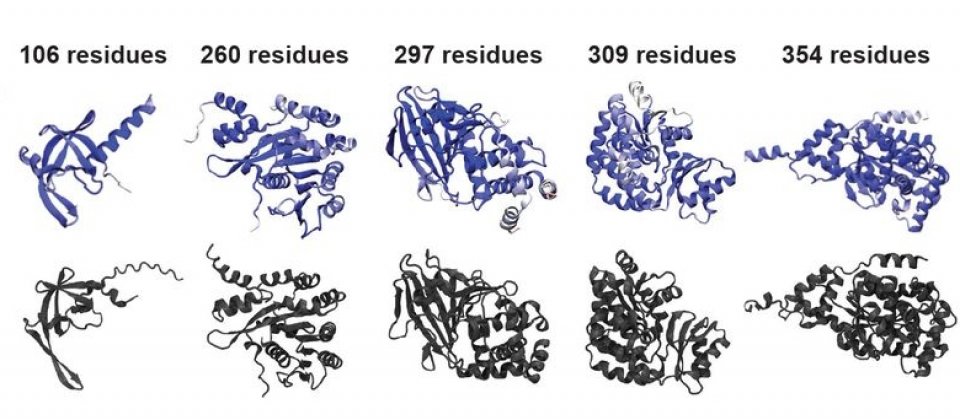
使用EvoDiff框架所訓練的模型,主要功能在於能生成符合現實且多樣化的新蛋白質,EvoDiff的設計能夠生成所有自然蛋白質中存在的結構、功能和序列特徵,也就是說,EvoDiff模型能夠在序列空間生成,在生物學和疾病中都有非常重要作用的無序蛋白質(Disordered Protein)。
EvoDiff框架的貢獻在於能夠在序列空間生成高品質蛋白質,打開了許多蛋白質工程和設計的可能性。微軟現已將EvoDiff的程式碼在GitHub上開源,供其他研究人員使用。
熱門新聞
2026-02-11
2026-02-11
2026-02-09
2026-02-10
2026-02-10
2026-02-06
2026-02-10